Regresiones para Respuestas Discretas
Contenido
17. Regresiones para Respuestas Discretas¶
En esta sesión vamos a analizar una generalización del modelo de regresion lineal que permite modelar un conjunto de datos, en que la variable a explicar es discreta y las variables explicativas son numéricas o categóricas. Dos ejemplos de ello son los modelos de regresión logística y regresión de Poisson, que estimaremos en R con ayuda de la función glm.
17.1. Regresión Logística¶
En este caso la variable a explicar es binaria y las variables explicativas son numéricas o categóricas.
Formalmente:
Sea \(y \in \{0,1\}\) la variable a explicar y \(\textbf{x} \in \mathbb{R}^k\) las variables explicativas.
Dos cambios respecto de la regresión lineal:
El primero es que \(y |\textbf{x}\) se modela como una v.a. con distribución \(\textbf{Bernoulli}\) en lugar de una Normal, es decir:
dónde
Y lo segundo es que, al igual que en la regresión lineal, se considera una combinación lineal de las variables explicativas, pero esta combinación lineal es pasada como parámetro a una función \(\mu\) que toma valores en el intervalo \([0,1]\), de manera que:
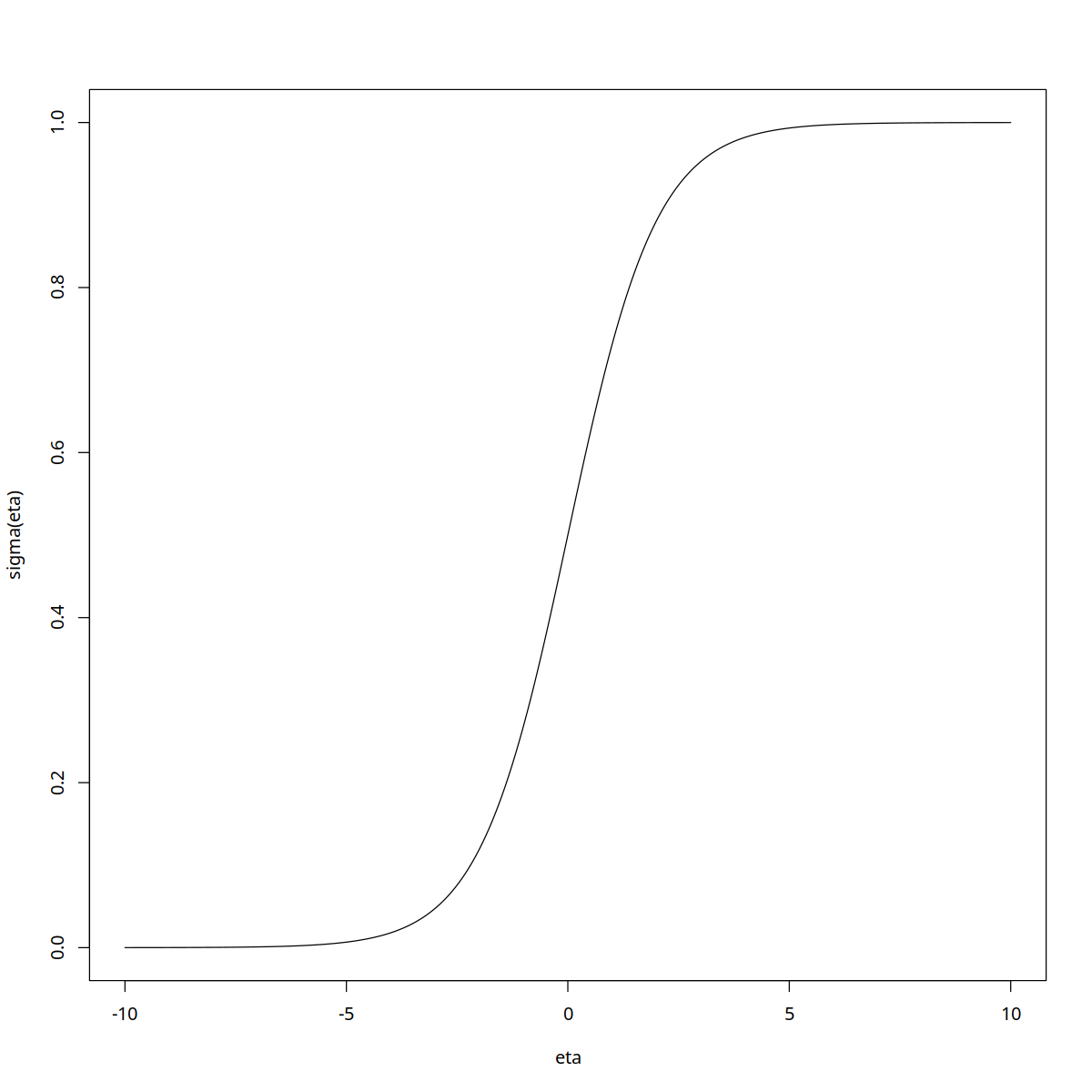
dónde \(\sigma\) es la función sigmoidal definida por:
y su inversa conocida también como \(\textbf{logística}\) o \(\textbf{logit}\) es:
entonces se cumple que
Asi, se obtiene el modelo:
denominado regresión logística, por su similitud con la regresión lineal, aunque es una forma de clasificación mas que una regresión.
El coeficiente \(\frac{p}{1-p}\) se denomina suele denominar riesgo (odd en inglés) pues es la tasa entre las probabilidades de respuesta “positiva” y “negativa”.
suppressMessages(library(ggplot2))
suppressMessages(library(grid))
suppressMessages(library(grDevices))
#gráfica de la función sigmoide
options(repr.plot.width=10, repr.plot.height=10)
eta = seq(-10,10,by=0.01)
sigma <- function(x){return( exp(x)/(exp(x)+1))}
plot(eta,sigma(eta),type="l")
17.1.1. Estimación de parámetros \(\beta\)¶
Los parámetros \(\beta\) se pueden estimar por máxima verosimilitud. En efecto, sea la muestra \(\{(y_i,\textbf{x}_i), i=1,\cdots,n\}\), se tiene:
A partir de esta función de verosimilitud no es posible obtener de manera analítica los estimadores máximo verosímiles de \(\beta\). Se requiere utilizar algún algoritmo de aproximación como Newton-Raphson para las raices del gradiente de \(\log \mathcal{L}(\beta)\) o utilizar directamente el algoritmo del gradiente descendente.
En el caso de Newton-Raphson las ecuaciones de aproximación quedan:
donde \(X \in M_{nxk}\) es la matriz de valores de las \(k\) variables explicativas en las \(n\) observaciones,
y \(W \in M_{nxn}\) es la matriz diagonal con:
Lo criterios de convergencia del método iterativo utilizado para estimar los parámetros pueden ser varios, pero en todos ellos la idea subyacente es que:
#Ejemplo de regresión logística para datos SAT
datos <- read.csv("datosSAT.csv",header=TRUE,sep=";")
summary(datos)
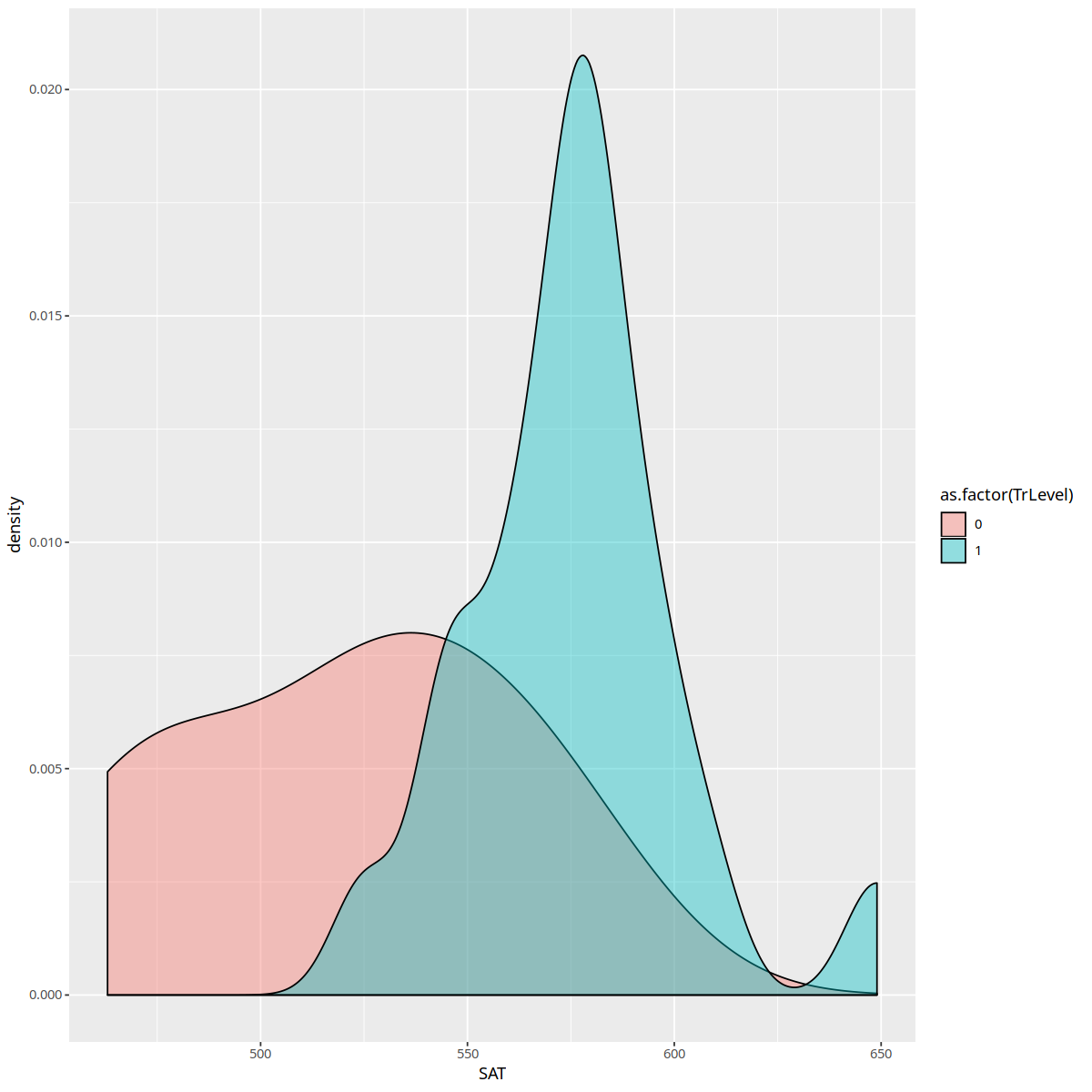
#TrLevel es el nivel alcanzado: 1 si pasó el curso 0 si no
#SAT es el puntaje obtenido en el test de Lenguaje
ggplot(datos, aes(SAT, fill = as.factor(TrLevel))) +
geom_density(alpha = .4)
TrLevel SAT
Min. :0.0 Min. :463.0
1st Qu.:0.0 1st Qu.:543.5
Median :1.0 Median :567.5
Mean :0.7 Mean :559.5
3rd Qu.:1.0 3rd Qu.:582.0
Max. :1.0 Max. :649.0
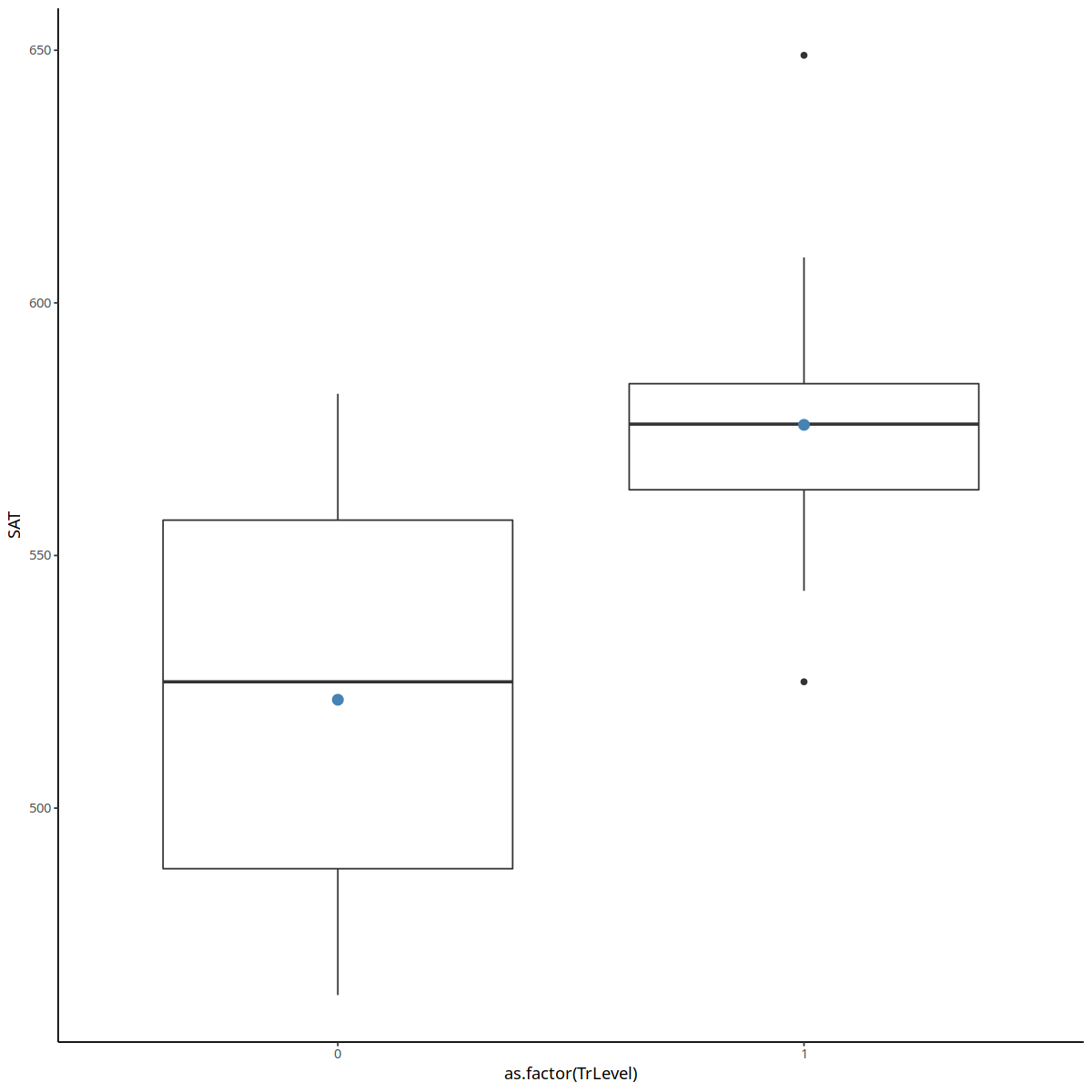
ggplot(datos, aes(x = as.factor(TrLevel), y = SAT)) +
geom_boxplot() +
stat_summary(fun = mean,
geom = "point",
size = 3,
color = "steelblue") +
theme_classic()
Warning message:
“Ignoring unknown parameters: fun”No summary function supplied, defaulting to `mean_se()
res<- glm(TrLevel ~ SAT, data=datos,family = binomial(link = "logit"))
summary(res)
Call:
glm(formula = TrLevel ~ SAT, family = binomial(link = "logit"),
data = datos)
Deviance Residuals:
Min 1Q Median 3Q Max
-2.2918 -0.1830 0.3662 0.5057 1.5301
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -31.65827 12.69071 -2.495 0.0126 *
SAT 0.05878 0.02296 2.560 0.0105 *
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 36.652 on 29 degrees of freedom
Residual deviance: 21.993 on 28 degrees of freedom
AIC: 25.993
Number of Fisher Scoring iterations: 5
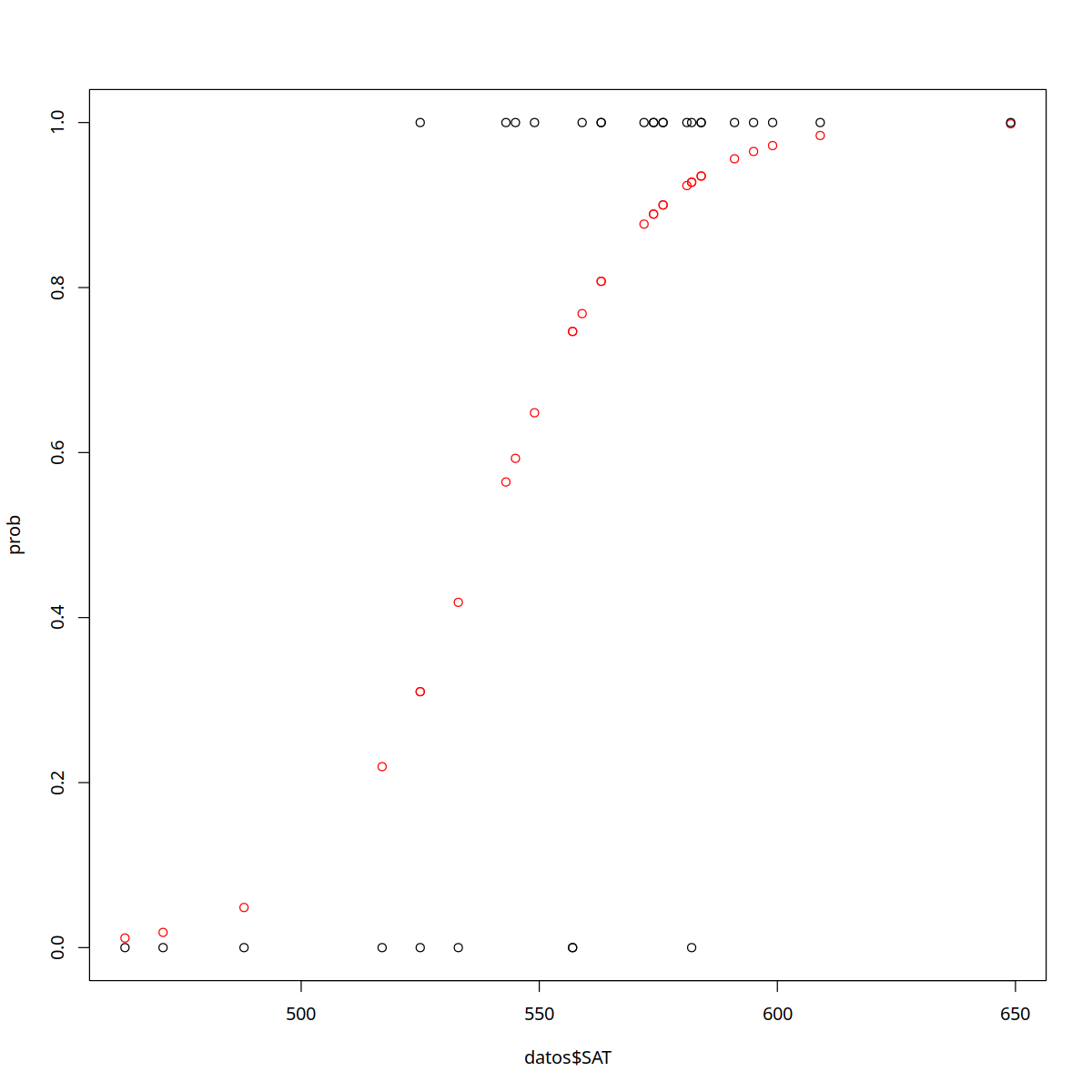
#graficamos los resultados
dat <-cbind(rep(1,30),datos$SAT)
prob <- sigma(dat%*%res$coefficients)
plot(datos$SAT,prob,col="red",ylim=c(0,1))
points(datos$SAT,datos$TrLevel)
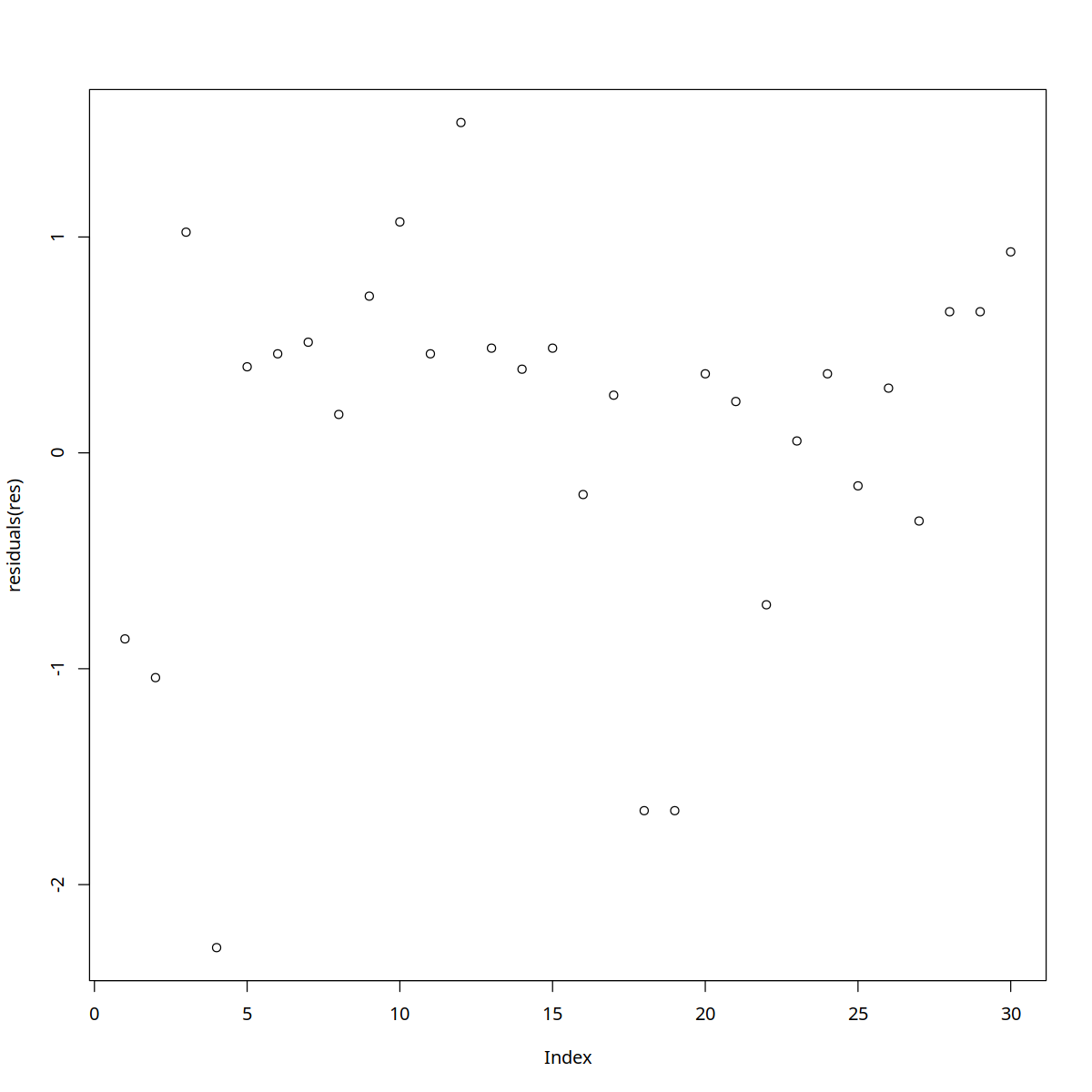
17.1.2. Evaluación del Ajuste del modelo¶
Cálculo de residuos
Si notamos $\( \hat{p}_i = \sigma(\hat{\beta}^T x_i)\)$
entonces se define el residuo en la verosimilitud correspondiente a la observación \(i\) en el modelo estimado como:
plot(residuals(res))
Coeficiente de información de Akaike, AIC
Recordemos que
donde k es el número de parámetros del modelo. Cuando se comparan modelos se escoge aquel con menor AIC.
Error de clasificación: Matriz de confusión
La matriz de confusión permite analizar los errores de clasificación:
Error tipo I o “falso positivo”: caso predicho con valor 1, cuando se observa 0
Error tipo II o “falso negativo”: caso predicho con valor 0 cuando se observa 1
tab <- table(prob>0.5, as.factor(datos$TrLevel))
print(tab)
error <- (tab[2,1]+tab[1,2])/sum(tab)
errorI <- tab[2,1]/(tab[2,1]+tab[2,2])
errorII <- tab[1,2]/(tab[1,1]+tab[1,2])
print(c(errorI,errorII, error))
0 1
FALSE 6 1
TRUE 3 20
[1] 0.1304348 0.1428571 0.1333333
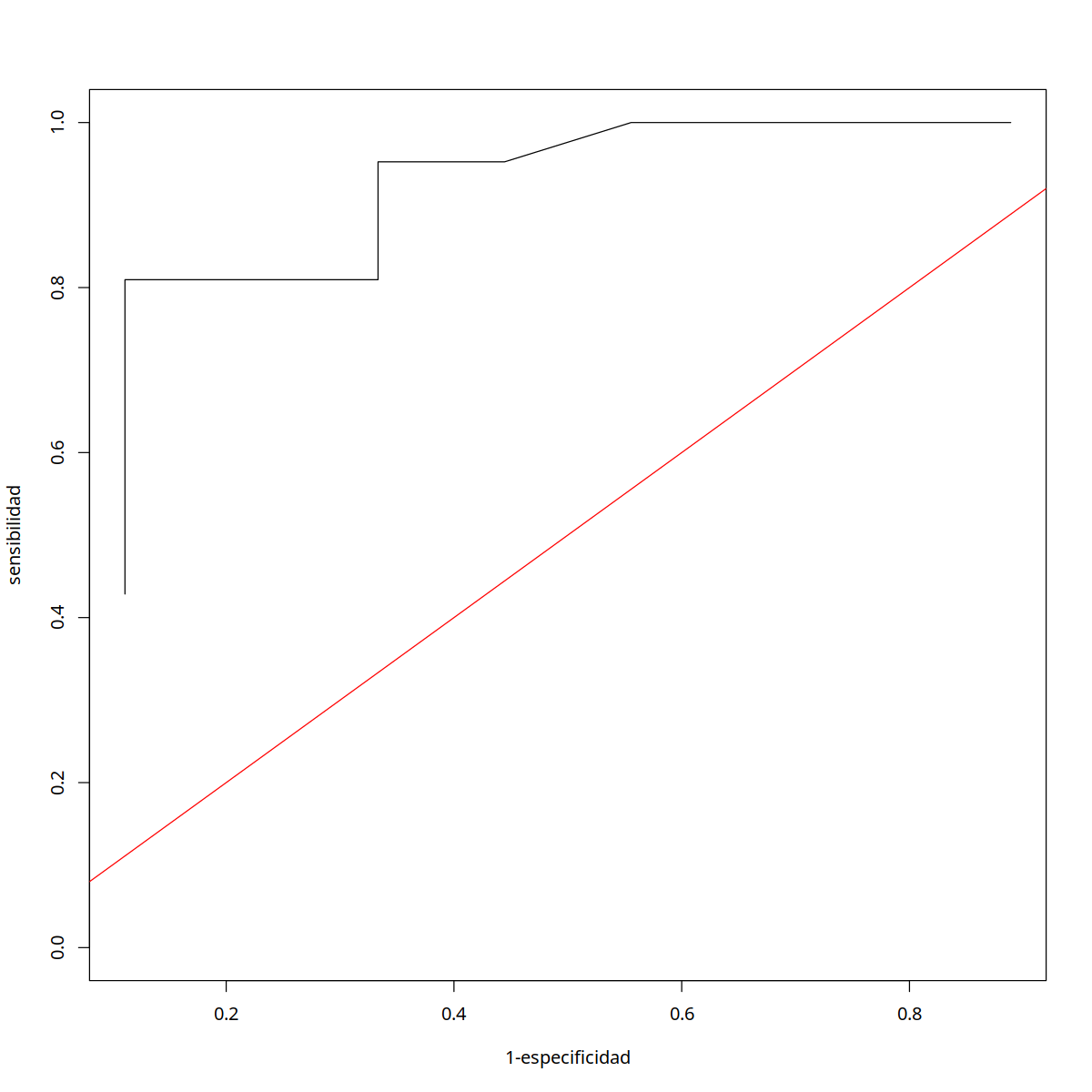
Cuantificando Habilidad Predictiva: Curva ROC
Este análisis considera variar el valor p(=0.5 por omisión) que guía la clasificación de los datos y se observa como varían la sensibilidad (tasa de verdaderos positivos sobre el total de positivos) en relación al inverso de la especificidad (tasa de verdaderos negativos sobre el total de negativos). Si el área bajo la curva ROC es cercana a 1 se trata de un buen clasificador, si es cercana a 0.5 corresponde a una clasificación aleatoria (igual probabilidad de positivo o negativo).
min(prob)
p1=matrix(0,nrow=19,ncol=3)
i=1
for(p in seq(min(prob),.95,.05)){
t1=table(prob>p,as.factor(datos$TrLevel))
p1[i,]=c(p,(t1[2,2])/sum(t1[,2]),(t1[1,1])/sum(t1[,1]))
i=i+1
}
print(p1)
plot(1-p1[,3],p1[,2],type="l",xlab="1-especificidad",ylab="sensibilidad",ylim=c(0,1))
lines(c(0,1),c(0,1),col="red")
[,1] [,2] [,3]
[1,] 0.01161768 1.0000000 0.1111111
[2,] 0.06161768 1.0000000 0.3333333
[3,] 0.11161768 1.0000000 0.3333333
[4,] 0.16161768 1.0000000 0.3333333
[5,] 0.21161768 1.0000000 0.3333333
[6,] 0.26161768 1.0000000 0.4444444
[7,] 0.31161768 0.9523810 0.5555556
[8,] 0.36161768 0.9523810 0.5555556
[9,] 0.41161768 0.9523810 0.5555556
[10,] 0.46161768 0.9523810 0.6666667
[11,] 0.51161768 0.9523810 0.6666667
[12,] 0.56161768 0.9523810 0.6666667
[13,] 0.61161768 0.8571429 0.6666667
[14,] 0.66161768 0.8095238 0.6666667
[15,] 0.71161768 0.8095238 0.6666667
[16,] 0.76161768 0.8095238 0.8888889
[17,] 0.81161768 0.6666667 0.8888889
[18,] 0.86161768 0.6666667 0.8888889
[19,] 0.91161768 0.4285714 0.8888889
# Multiple plot function
#
# ggplot objects can be passed in ..., or to plotlist (as a list of ggplot objects)
# - cols: Number of columns in layout
# - layout: A matrix specifying the layout. If present, 'cols' is ignored.
#
# If the layout is something like matrix(c(1,2,3,3), nrow=2, byrow=TRUE),
# then plot 1 will go in the upper left, 2 will go in the upper right, and
# 3 will go all the way across the bottom.
#
multiplot <- function(..., plotlist=NULL, file, cols=1, layout=NULL) {
# Make a list from the ... arguments and plotlist
plots <- c(list(...), plotlist)
numPlots = length(plots)
# If layout is NULL, then use 'cols' to determine layout
if (is.null(layout)) {
# Make the panel
# ncol: Number of columns of plots
# nrow: Number of rows needed, calculated from # of cols
layout <- matrix(seq(1, cols * ceiling(numPlots/cols)),
ncol = cols, nrow = ceiling(numPlots/cols))
}
if (numPlots==1) {
print(plots[[1]])
} else {
# Set up the page
grid.newpage()
pushViewport(viewport(layout = grid.layout(nrow(layout), ncol(layout))))
# Make each plot, in the correct location
for (i in 1:numPlots) {
# Get the i,j matrix positions of the regions that contain this subplot
matchidx <- as.data.frame(which(layout == i, arr.ind = TRUE))
print(plots[[i]], vp = viewport(layout.pos.row = matchidx$row,
layout.pos.col = matchidx$col))
}
}
}
#Ejemplo de regresión logística para datos de PSU de estudiantes de primer año universitario
#y su relación con la estrategia de aprendizaje Elaborativa del estudiante (0, bajo nivel - 1 normal o alto nivel)
#obtenida en un test de diagnóstico
datos <- read.csv("DatosEjemplo.csv",header=TRUE,sep=";")
print(datos[1:10,])
summary(datos)
Elaborativo MAT LENG CIENCIA
1 0 429 573 443
2 0 572 534 497
3 1 631 692 611
4 0 713 654 694
5 1 591 523 537
6 0 610 530 558
7 1 619 628 409
8 0 596 628 625
9 1 644 466 523
10 0 644 628 591
Elaborativo MAT LENG CIENCIA
Min. :0.0000 Min. :429.0 Min. :442.0 Min. :371.0
1st Qu.:0.0000 1st Qu.:581.0 1st Qu.:530.0 1st Qu.:523.0
Median :1.0000 Median :605.0 Median :573.0 Median :570.0
Mean :0.6796 Mean :603.8 Mean :576.9 Mean :560.7
3rd Qu.:1.0000 3rd Qu.:636.0 3rd Qu.:620.0 3rd Qu.:606.0
Max. :1.0000 Max. :725.0 Max. :716.0 Max. :694.0
dim(datos)
- 103
- 4
options(repr.plot.width=20, repr.plot.height=6)
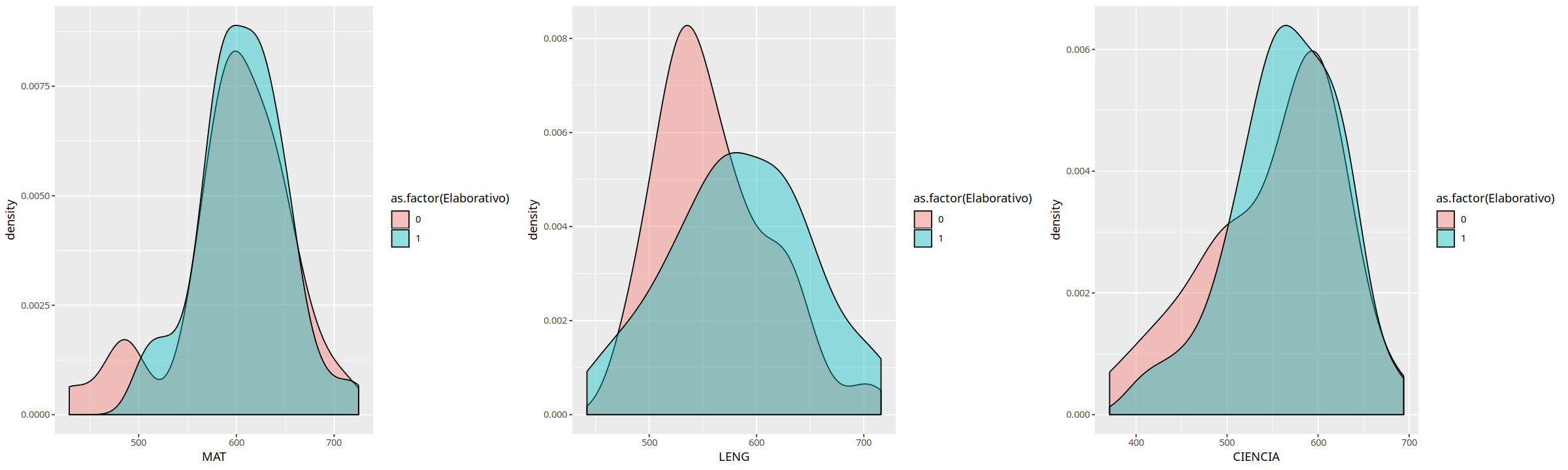
f1 <- ggplot(datos, aes(MAT, fill = as.factor(Elaborativo))) +
geom_density(alpha = .4)
f2 <- ggplot(datos, aes(LENG, fill = as.factor(Elaborativo))) +
geom_density(alpha = .4)
f3 <- ggplot(datos, aes(CIENCIA, fill = as.factor(Elaborativo))) +
geom_density(alpha = .4)
multiplot(f1,f2,f3, cols=3)
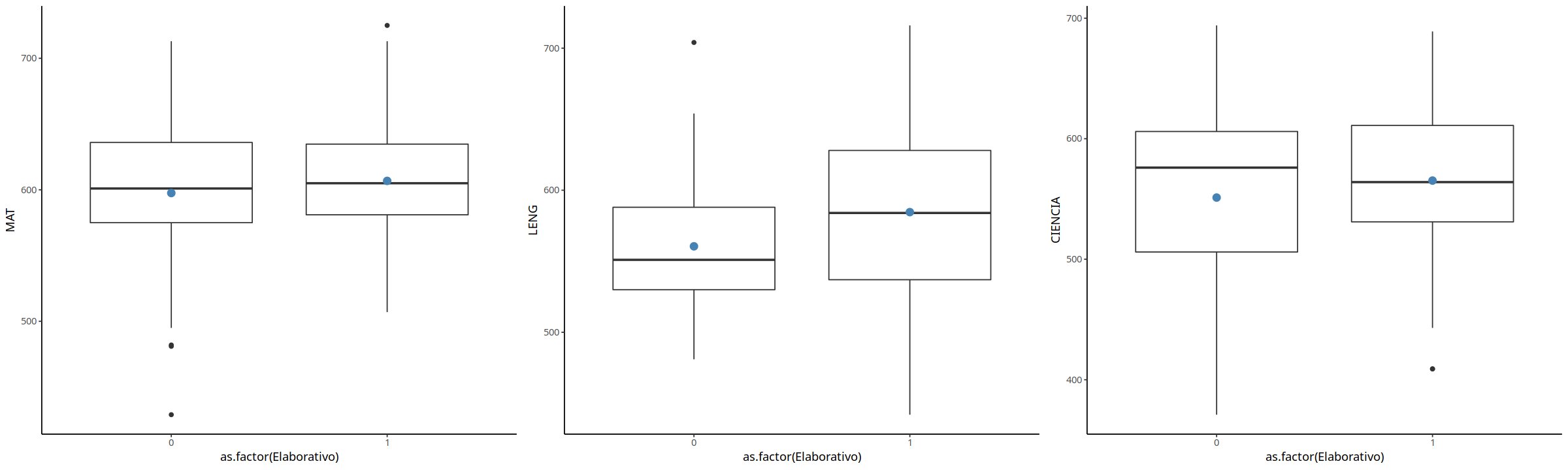
f1 <- ggplot(datos, aes(x = as.factor(Elaborativo), y = MAT)) +
geom_boxplot() +
stat_summary(fun = mean,
geom = "point",
size = 3,
color = "steelblue") +
theme_classic()
f2<- ggplot(datos, aes(x = as.factor(Elaborativo), y = LENG)) +
geom_boxplot() +
stat_summary(fun = mean,
geom = "point",
size = 3,
color = "steelblue") +
theme_classic()
f3 <- ggplot(datos, aes(x = as.factor(Elaborativo), y = CIENCIA)) +
geom_boxplot() +
stat_summary(fun = mean,
geom = "point",
size = 3,
color = "steelblue") +
theme_classic()
multiplot(f1,f2,f3, cols=3)
Warning message:
“Ignoring unknown parameters: fun”Warning message:
“Ignoring unknown parameters: fun”Warning message:
“Ignoring unknown parameters: fun”No summary function supplied, defaulting to `mean_se()
No summary function supplied, defaulting to `mean_se()
No summary function supplied, defaulting to `mean_se()
## análisis con las 3 variables
res0<- glm(Elaborativo ~ MAT+LENG+CIENCIA, data=datos,family = binomial(link = "logit"))
summary(res0)
Call:
glm(formula = Elaborativo ~ MAT + LENG + CIENCIA, family = binomial(link = "logit"),
data = datos)
Deviance Residuals:
Min 1Q Median 3Q Max
-1.9029 -1.3534 0.7578 0.8999 1.1856
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -3.4698760 2.8740756 -1.207 0.227
MAT 0.0009539 0.0055064 0.173 0.862
LENG 0.0062689 0.0041219 1.521 0.128
CIENCIA 0.0001063 0.0045875 0.023 0.982
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 129.20 on 102 degrees of freedom
Residual deviance: 125.65 on 99 degrees of freedom
AIC: 133.65
Number of Fisher Scoring iterations: 4
print(res0$coefficients)
(Intercept) MAT LENG CIENCIA
-3.4698760404 0.0009539276 0.0062689095 0.0001062675
dat <-cbind(rep(1,103),datos$MAT,datos$LENG,datos$CIENCIA)
vec <- res0$coefficients
prob0 <- sigma(dat%*%vec)
tab <- table(prob0>0.5, as.factor(datos$Elaborativo))
print(tab)
error <- (tab[2,1]+tab[1,2])/sum(tab)
errorI <- tab[2,1]/(tab[2,1]+tab[2,2])
errorII <- tab[1,2]/(tab[1,1]+tab[1,2])
print(c(errorI,errorII, error))
0 1
FALSE 0 3
TRUE 33 67
[1] 0.3300000 1.0000000 0.3495146
options(repr.plot.width=8, repr.plot.height=8)
p0=matrix(0,nrow=8,ncol=3)
i=1
for(p in seq(min(prob0),.85,.05)){
t1=table(prob0>p,as.factor(datos$Elaborativo))
p0[i,]=c(p,(t1[2,2])/sum(t1[,2]),(t1[1,1])/sum(t1[,1]))
i=i+1
}
print(p0)
plot(1-p0[,3],p0[,2],type="l",xlab="1-especificidad",ylab="sensibilidad")
lines(c(0,1),c(0,1),col="red")
[,1] [,2] [,3]
[1,] 0.4952148 0.98571429 0.00000000
[2,] 0.5452148 0.90000000 0.06060606
[3,] 0.5952148 0.87142857 0.21212121
[4,] 0.6452148 0.70000000 0.54545455
[5,] 0.6952148 0.51428571 0.69696970
[6,] 0.7452148 0.30000000 0.81818182
[7,] 0.7952148 0.10000000 0.93939394
[8,] 0.8452148 0.02857143 1.00000000
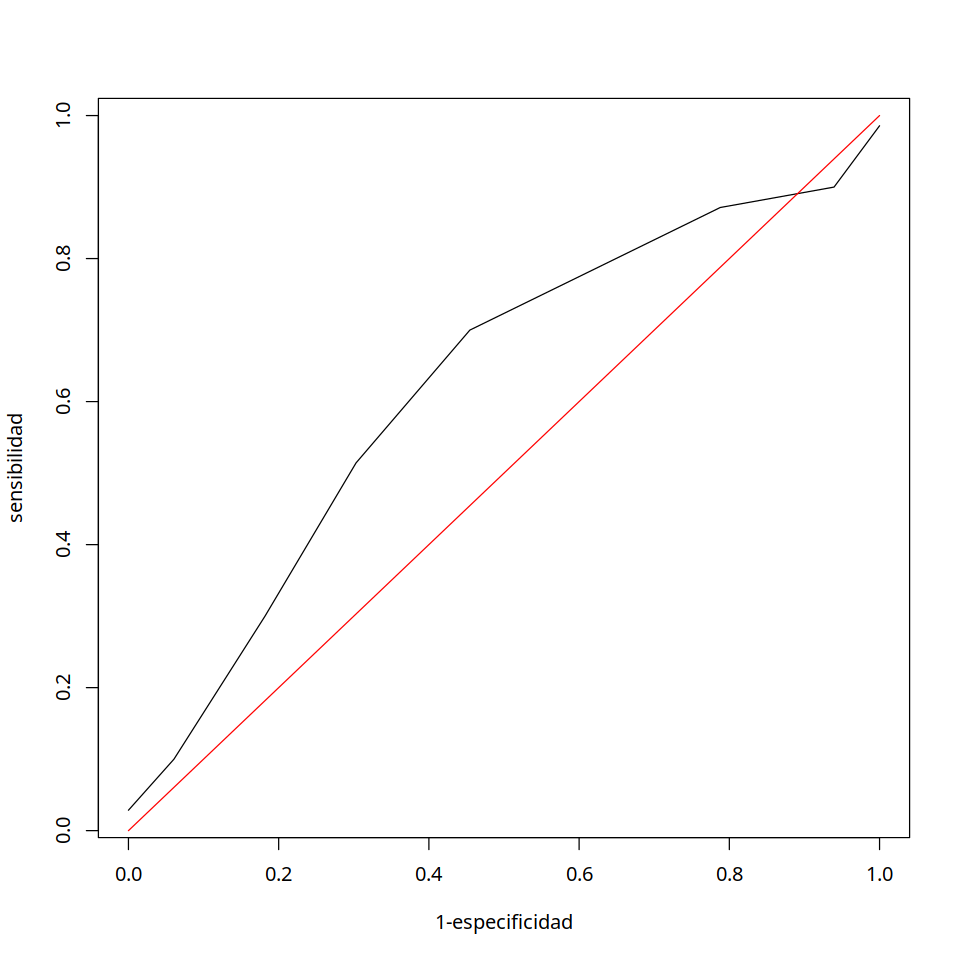
## análisis con 2 variables
res1<- glm(Elaborativo ~ MAT+LENG, data=datos,family = binomial(link = "logit"))
summary(res1)
Call:
glm(formula = Elaborativo ~ MAT + LENG, family = binomial(link = "logit"),
data = datos)
Deviance Residuals:
Min 1Q Median 3Q Max
-1.9033 -1.3510 0.7515 0.8978 1.1853
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -3.473364 2.870078 -1.210 0.226
MAT 0.001026 0.004539 0.226 0.821
LENG 0.006303 0.003856 1.635 0.102
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 129.20 on 102 degrees of freedom
Residual deviance: 125.65 on 100 degrees of freedom
AIC: 131.65
Number of Fisher Scoring iterations: 4
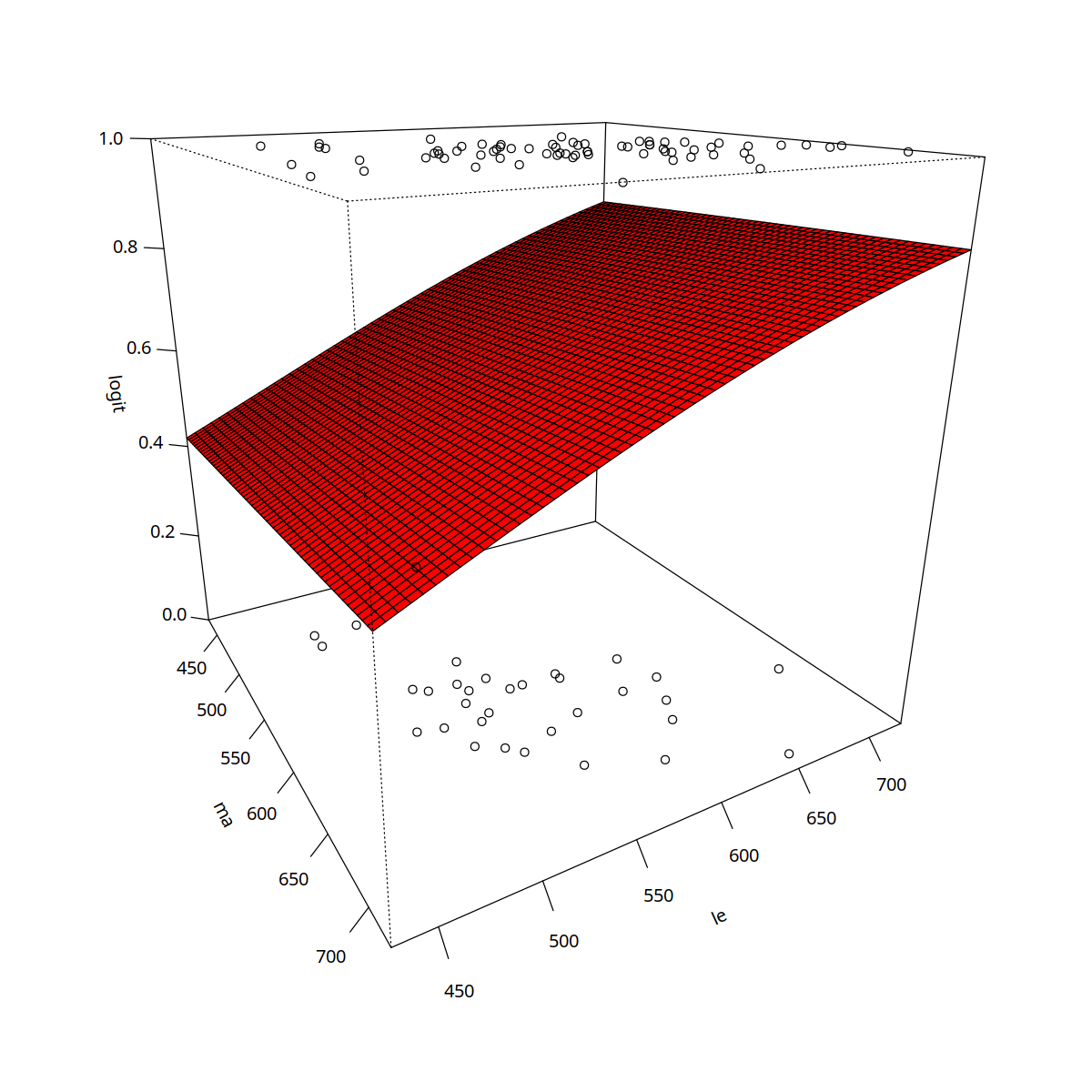
#graficamos los resultados
options(repr.plot.width=10, repr.plot.height=10)
ma <- seq(429,726,by=5)
le <- seq(429,726,by=5)
dens <- function(x,y) sigma(res1$coefficients[1] + x*res1$coefficients[2] + y*res1$coefficients[3] )
z <- outer(ma,le,dens)
pmat<- persp(ma,le,z,ticktype = "detailed",zlab="logit",zlim=c(0,1), phi=20,theta=60,col="red")
points(trans3d(datos$MAT,datos$LENG,datos$Elaborativo,pmat=pmat))
dat <-cbind(rep(1,103),datos$MAT,datos$LENG)
prob1 <- sigma(dat%*%res1$coefficients)
tab <- table(prob1>0.5, as.factor(datos$Elaborativo))
print(tab)
error <- (tab[2,1]+tab[1,2])/sum(tab)
errorI <- tab[2,1]/(tab[2,1]+tab[2,2])
errorII <- tab[1,2]/(tab[1,1]+tab[1,2])
print(c(errorI,errorII, error))
0 1
FALSE 0 3
TRUE 33 67
[1] 0.3300000 1.0000000 0.3495146
options(repr.plot.width=8, repr.plot.height=8)
p1=matrix(0,nrow=8,ncol=3)
i=1
for(p in seq(min(prob1),.85,.05)){
t1=table(prob1>p,as.factor(datos$Elaborativo))
p1[i,]=c(p,(t1[2,2])/sum(t1[,2]),(t1[1,1])/sum(t1[,1]))
i=i+1
}
print(p1)
plot(1-p1[,3],p1[,2],type="l",xlab="1-especificidad",ylab="sensibilidad")
lines(c(0,1),c(0,1),col="red")
[,1] [,2] [,3]
[1,] 0.4953638 0.98571429 0.00000000
[2,] 0.5453638 0.88571429 0.06060606
[3,] 0.5953638 0.87142857 0.21212121
[4,] 0.6453638 0.71428571 0.54545455
[5,] 0.6953638 0.51428571 0.69696970
[6,] 0.7453638 0.30000000 0.81818182
[7,] 0.7953638 0.10000000 0.93939394
[8,] 0.8453638 0.02857143 1.00000000
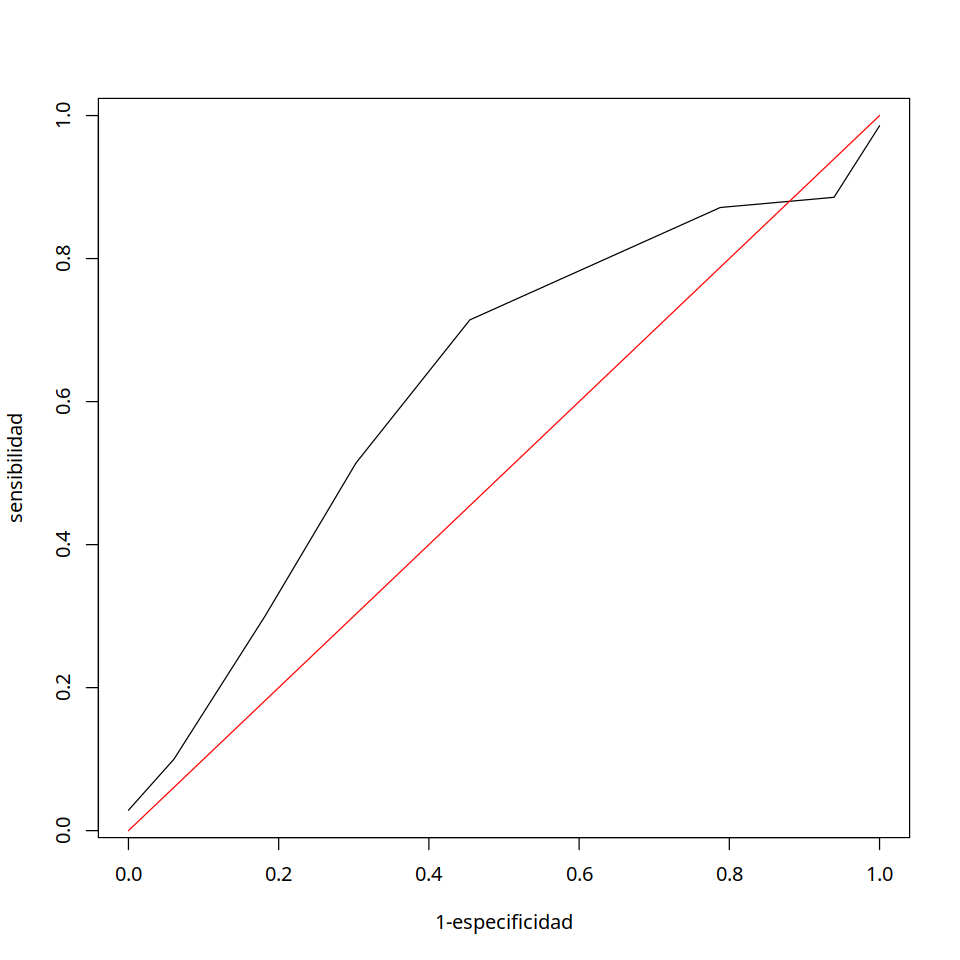
res2<- glm(Elaborativo ~ LENG, data=datos,family = binomial(link = "logit"))
summary(res2)
Call:
glm(formula = Elaborativo ~ LENG, family = binomial(link = "logit"),
data = datos)
Deviance Residuals:
Min 1Q Median 3Q Max
-1.8979 -1.3454 0.7518 0.8943 1.2238
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -3.025041 2.071847 -1.460 0.1443
LENG 0.006599 0.003628 1.819 0.0689 .
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 129.2 on 102 degrees of freedom
Residual deviance: 125.7 on 101 degrees of freedom
AIC: 129.7
Number of Fisher Scoring iterations: 4
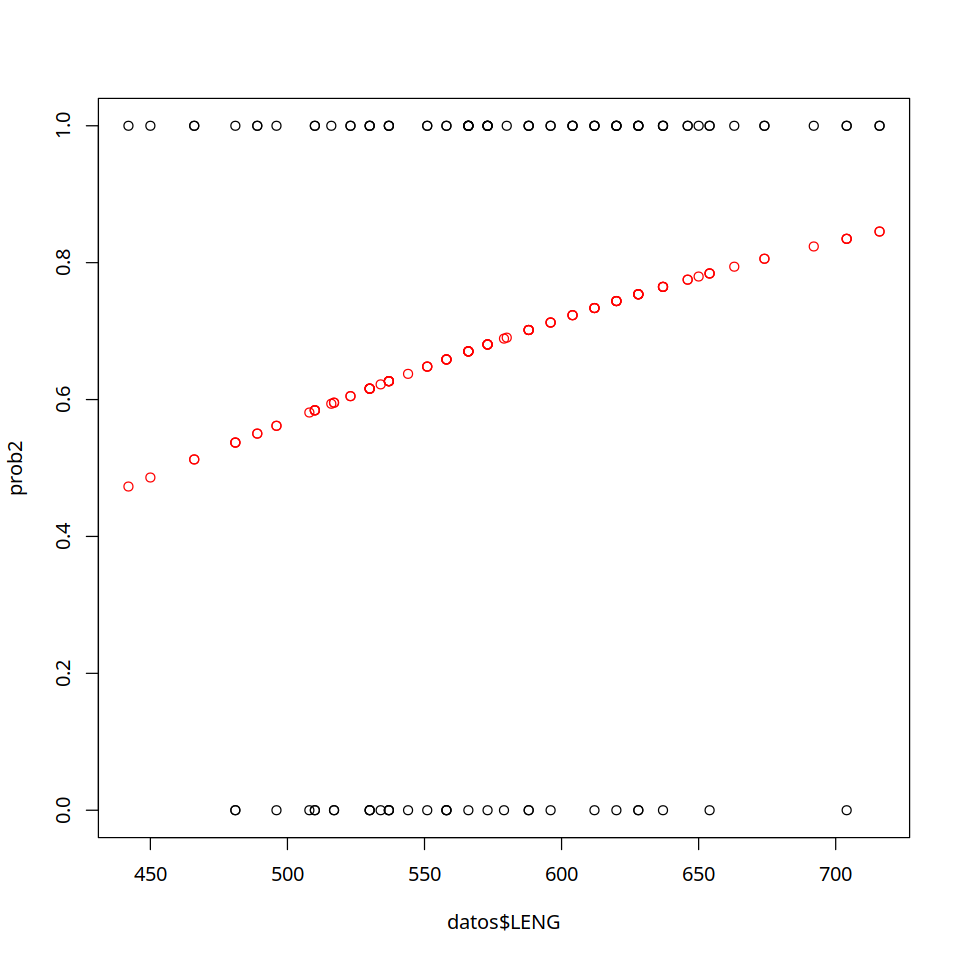
#graficamos los resultados
dat <-cbind(rep(1,103),datos$LENG)
prob2 <- sigma(dat%*%res2$coefficients)
plot(datos$LENG,prob2,col="red",ylim=c(0,1))
points(datos$LENG,datos$Elaborativo)
tab <- table(prob2>0.5, as.factor(datos$Elaborativo))
print(tab)
error <- (tab[2,1]+tab[1,2])/sum(tab)
errorI <- tab[2,1]/(tab[2,1]+tab[2,2])
errorII <- tab[1,2]/(tab[1,1]+tab[1,2])
print(c(errorI,errorII, error))
0 1
FALSE 0 2
TRUE 33 68
[1] 0.3267327 1.0000000 0.3398058
print(min(prob2))
[1] 0.4729352
options(repr.plot.width=16, repr.plot.height=6)
p2=matrix(0,nrow=8,ncol=3)
i=1
for(p in seq(min(prob2),.85,.05)){
t2=table(prob2>p,as.factor(datos$Elaborativo))
p2[i,]=c(p,(t2[2,2])/sum(t2[,2]),(t2[1,1])/sum(t2[,1]))
i=i+1
}
print(p2)
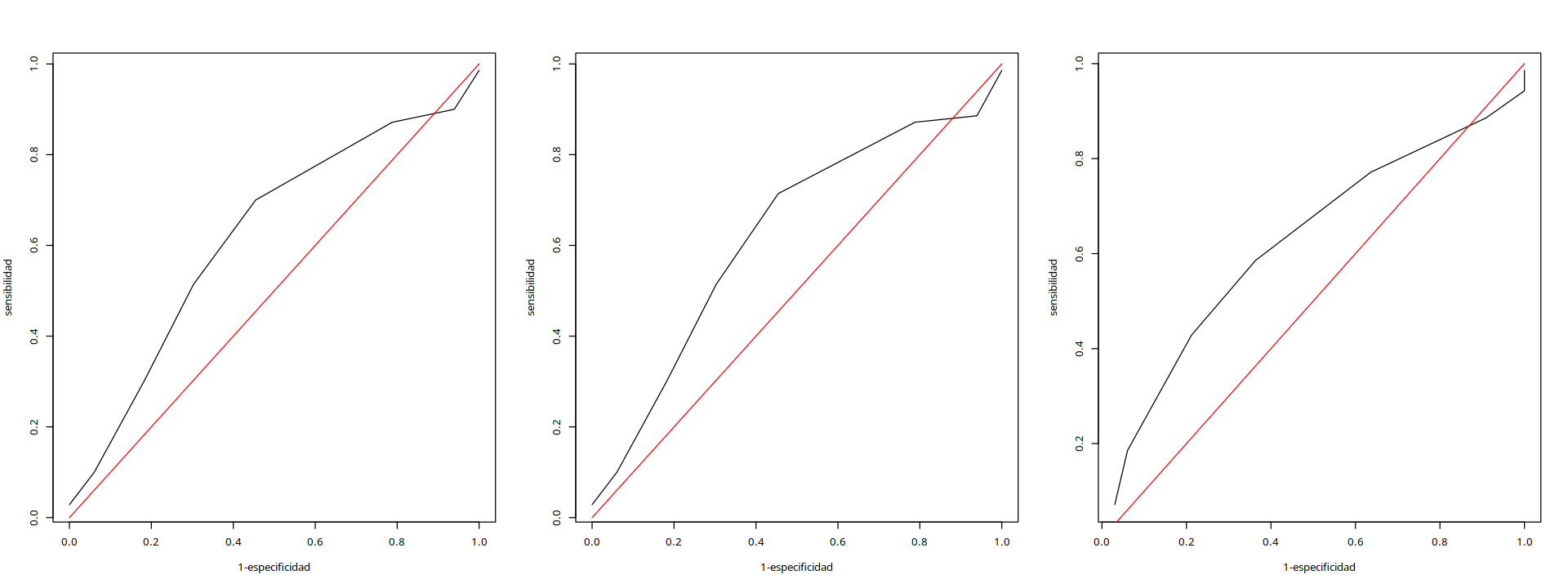
par(mfrow=c(1,3))
plot(1-p0[,3],p0[,2],type="l",xlab="1-especificidad",ylab="sensibilidad")
lines(c(0,1),c(0,1),col="red")
plot(1-p1[,3],p1[,2],type="l",xlab="1-especificidad",ylab="sensibilidad")
lines(c(0,1),c(0,1),col="red")
plot(1-p2[,3],p2[,2],type="l",xlab="1-especificidad",ylab="sensibilidad")
lines(c(0,1),c(0,1),col="red")
[,1] [,2] [,3]
[1,] 0.4729352 0.98571429 0.00000000
[2,] 0.5229352 0.94285714 0.00000000
[3,] 0.5729352 0.88571429 0.09090909
[4,] 0.6229352 0.77142857 0.36363636
[5,] 0.6729352 0.58571429 0.63636364
[6,] 0.7229352 0.42857143 0.78787879
[7,] 0.7729352 0.18571429 0.93939394
[8,] 0.8229352 0.07142857 0.96969697
17.2. Regresión de Poisson¶
En este caso la variable a explicar es discreta, mas precisamente es una variable de conteo o a valores en \(\it N\) y las variables explicativas son numéricas o categóricas.
Formalmente:
Sea \(y \in \{0,1,2, \cdots\}\) la variable a explicar y \(\textbf{x} \in \mathbb{R}^k\) las variables explicativas.
Entonces \(y\mid \textbf{x}\) se modela como una v.a. con distribución \(\textbf{Poisson}\), es decir:
dónde, al igual que en la regresión lineal, se considera una combinación lineal de las variables explicativas, pero esta combinación lineal es pasada como parámetro a una función \( \lambda\) que toma valores positivos:
Así,
Notar que en este caso, se tiene:
17.2.1. Estimación de parámetros \(\beta\)¶
Los parámetros \(\beta\) se pueden estimar por máxima verosimilitud. En efecto, sea la muestra \(\{(y_i,\textbf{x}_i), i=1,\cdots,n\}\), se tiene:
A partir de esta función de verosimilitud no es posible obtener de manera analítica los estimadores máximo verosímiles de \(\beta\). Utilizando Newton-Raphson para aproximar las raices del gradiente de \(\log \mathcal{L}(\beta)\) se obtiene:
donde \(X \in M_{nxk}\) es la matriz de valores de las \(k\) variables explicativas en las \(n\) observaciones,
y \(W \in M_{nxn}\) es la matriz diagonal con:
Lo criterios de convergencia del método iterativo utilizado para estimar los parámetros pueden ser varios, pero en todos ellos la idea subyacente es que
#ejemplo de regresión de Poisson que considera datos simulados sobre 200 estudiantes de EM
#la variable a explicar es el número de premios obtenidos y las variables explicativas
#son sus puntajes en el examen final de matemáticas, y el tipo de programa en que
#están matriculados: general, vocacional o académico.
require(ggplot2)
options(repr.plot.width=10, repr.plot.height=10)
p <- read.csv("https://stats.idre.ucla.edu/stat/data/poisson_sim.csv")
p <- within(p, {
prog <- factor(prog, levels=1:3, labels=c("General", "Academic",
"Vocational"))
id <- factor(id)
})
print(p[1:20,])
summary(p)
id num_awards prog math
1 45 0 Vocational 41
2 108 0 General 41
3 15 0 Vocational 44
4 67 0 Vocational 42
5 153 0 Vocational 40
6 51 0 General 42
7 164 0 Vocational 46
8 133 0 Vocational 40
9 2 0 Vocational 33
10 53 0 Vocational 46
11 1 0 Vocational 40
12 128 0 Academic 38
13 16 0 Vocational 44
14 106 0 Vocational 37
15 89 0 Vocational 40
16 134 0 General 39
17 19 0 General 43
18 145 0 Vocational 38
19 11 0 Academic 45
20 117 0 Vocational 39
id num_awards prog math
1 : 1 Min. :0.00 General : 45 Min. :33.00
2 : 1 1st Qu.:0.00 Academic :105 1st Qu.:45.00
3 : 1 Median :0.00 Vocational: 50 Median :52.00
4 : 1 Mean :0.63 Mean :52.65
5 : 1 3rd Qu.:1.00 3rd Qu.:59.00
6 : 1 Max. :6.00 Max. :75.00
(Other):194
with(p, tapply(num_awards, prog, function(x) {
sprintf("M (SD) = %1.2f (%1.2f)", mean(x), sd(x))
}))
- General
- 'M (SD) = 0.20 (0.40)'
- Academic
- 'M (SD) = 1.00 (1.28)'
- Vocational
- 'M (SD) = 0.24 (0.52)'
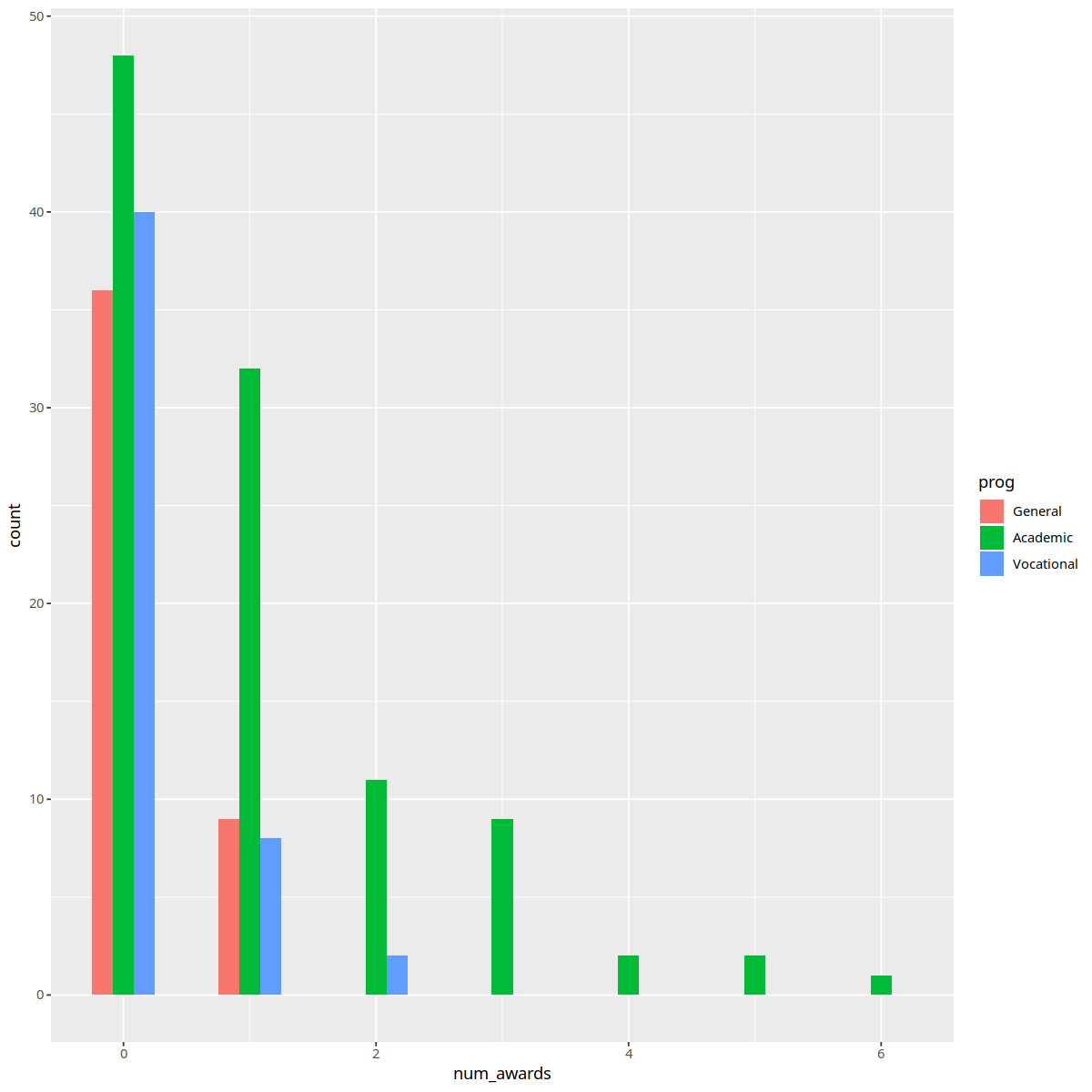
ggplot(p, aes(num_awards, fill = prog)) +
geom_histogram(binwidth=.5, position="dodge")
summary(m1 <- glm(num_awards ~ prog + math, family="poisson", data=p))
Call:
glm(formula = num_awards ~ prog + math, family = "poisson", data = p)
Deviance Residuals:
Min 1Q Median 3Q Max
-2.2043 -0.8436 -0.5106 0.2558 2.6796
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -5.24712 0.65845 -7.969 1.60e-15 ***
progAcademic 1.08386 0.35825 3.025 0.00248 **
progVocational 0.36981 0.44107 0.838 0.40179
math 0.07015 0.01060 6.619 3.63e-11 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
(Dispersion parameter for poisson family taken to be 1)
Null deviance: 287.67 on 199 degrees of freedom
Residual deviance: 189.45 on 196 degrees of freedom
AIC: 373.5
Number of Fisher Scoring iterations: 6
with(m1, cbind(res.deviance = deviance, df = df.residual,
p = pchisq(deviance, df.residual, lower.tail=FALSE)))
| res.deviance | df | p |
|---|---|---|
| 189.4496 | 196 | 0.6182274 |
## calculate and store predicted values
p$phat <- predict(m1, type="response")
## order by program and then by math
p <- p[with(p, order(prog, math)), ]
## create the plot
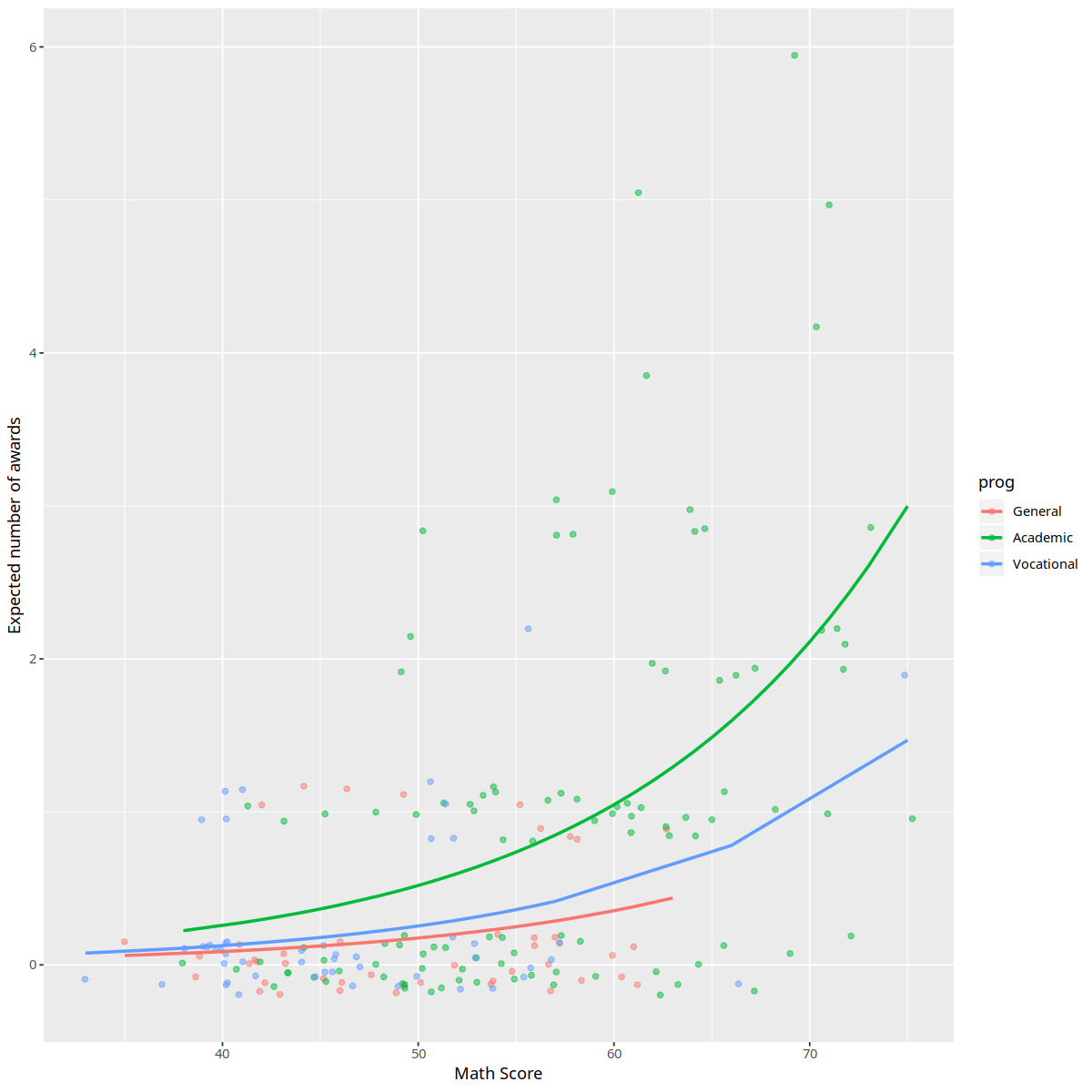
ggplot(p, aes(x = math, y = phat, colour = prog)) +
geom_point(aes(y = num_awards), alpha=.5, position=position_jitter(h=.2)) +
geom_line(size = 1) +
labs(x = "Math Score", y = "Expected number of awards")